Des bactéries impliquées dans le démantèlement des armes chimiques ?
Posté : 17 décembre 2020 / Mis-à-jour : 7 avril 2024
Temps de lecture : 7 minutes
De nombreuses bactéries sont capables de survivre et de se développer dans des environnements pollués. Certaines sont même capables de dégrader ces polluants et ainsi participer à la bioréhabilitation de sites pollués. Les polluants dégradés par les bactéries peuvent être d’origines industrielles mais aussi militaires comme les armes chimiques. Ces armes utilisent un composé chimique dans le but de blesser ou de tuer. Le gaz moutarde est un exemple d’arme chimique produite durant la première guerre mondiale. Cette arme chimique est encore trouvée dans l’environnement ce qui a conduit des scientifiques à étudier des bactéries capables de dégrader ce polluant [1].
Le gaz moutarde, une arme chimique encore d’actualité
Malgré son nom, le gaz moutarde n’est pas un gaz mais plutôt un liquide visqueux. Il est très soluble dans les graisses et pénètre facilement à travers la peau. Le gaz moutarde est responsable de sévères brulures et d’irritations au niveau de la peau, des yeux et du système respiratoire.
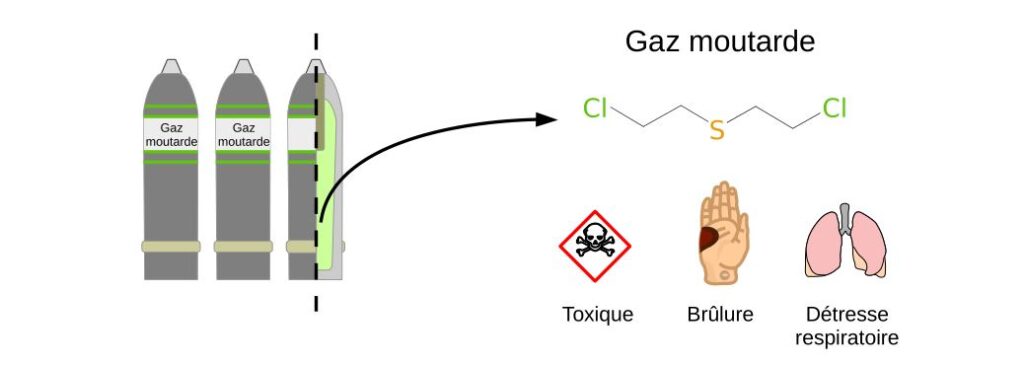
Cette molécule, et ses dérivés, endommagent également l’ADN conduisant à la mort cellulaire ou à des cancers. En plus d’être toxique pour les humains, le gaz moutarde est également dangereux pour l’environnement [2].
Malgré l’interdiction de son emploi par la convention sur les armes chimiques en 1997, le gaz moutarde est encore retrouvé dans l’environnement. Récemment, il a été utilisé en Syrie, lors de la guerre civile et par des groupes terroristes [3]. Des stocks d’armes n’ayant pas été détruits sont une autre source de gaz moutarde dans l’environnement. Par exemple, des stocks, datant de la seconde guerre mondiale, ont été coulés dans la mer Baltique [4 ; 5]. Malgré son séjour dans les fonds marins, ce polluant reste stable et cause des blessures aux pécheurs qui remontent à la surface des munitions endommagées.

Actuellement, des techniques physiques sont utilisées pour le démantèlement des stocks de gaz moutarde. Des scientifiques travaillent à mettre en place de nouvelles méthodes plus efficace et écologiques pour éliminer ce polluant à l’aide de bactéries [6].

Dégradation enzymatique du gaz moutarde
Certaines bactéries produisent une enzyme appelée DhaA qui est capable de dégrader le gaz moutarde ainsi que d’autres polluants contenant du chlore [7]. Une enzyme est un catalyseur chimique, c’est-à-dire une substance capable d’accélérer la vitesse d’une réaction chimique. L’enzyme DhaA est capable d’accélérer le retrait d’un atome de chlore présent sur des molécules organiques (à base de carbone) telle que le gaz moutarde.

L’utilisation d’enzymes présente plusieurs avantages pour la bioréhabilitation comme le fait qu’il s’agit d’un traitement non toxique, non abrasif et qu’il est réalisé dans des conditions normales de pressions et températures. De plus les enzymes sont biodégradables dans l’environnement et ne risquent donc pas de polluer l’environnement.
L’enzyme DhaA est produite notamment par la bactérie Rhodococcus rhodochrous NCIMB 13064 qui a été isolée d’un site industriel pollué [8]. Cette enzyme est synthétisée à l’intérieur de la bactérie (cytoplasme). Pour dépolluer un site contaminé, il est possible d’utiliser directement cette bactérie ou alors seulement l’enzyme qui a été purifiée à partir de la bactérie.

Pour obtenir l’enzyme DhaA purifiée, il faut cultiver la bactérie en grande quantité puis séparer cette molécule des autres présentes à l’intérieur de la bactérie. Pour cela, il faut lyser (détruire la membrane) de la bactérie pour libérer les molécules qu’elle contient. Lorsque l’on lyse une bactérie, on obtient un mélange de toutes les molécules contenues à l’intérieur de celle-ci. On peut dans ce cas parler de chercher « une aiguille dans une botte de foin » pour cette enzyme présente avec toutes les autres molécules produite par cette bactérie. Des étapes de purifications successives vont permettre de garder cette enzyme tout en éliminant les autres molécules issues de la bactérie.

La production et la purification d’une enzyme en grande quantité est une technique pouvant être longue et couteuse. Un autre problème est qu’une fois produite cette enzyme est peu stable. Cela signifie qu’une fois purifiée, elle perd rapidement son efficacité jusqu’à devenir inactive.
Une nouvelle approche pour produire l’enzyme DhaA
Une équipe de recherche chinoise propose une nouvelle méthode pour utiliser l’enzyme DhaA. Pour cela ils tirent partie d’une structure particulière produite par certaines bactéries : la spore [9]. Il s’agit d’une structure permettant de protéger la cellule lorsque les conditions de l’environnement deviennent néfastes pour la bactérie.

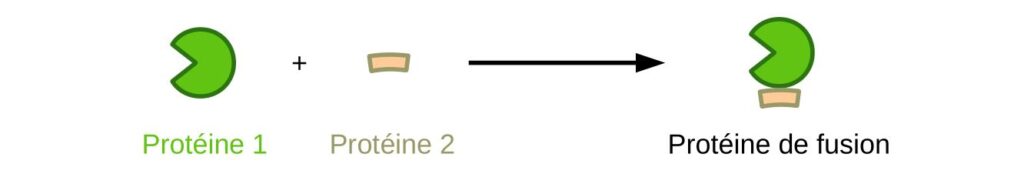
La stratégie proposée par ces scientifiques est de modifier l’une des protéines présente sur la spore pour la fusionner avec l’enzyme DhaA. Ainsi cette enzyme sera exposée sur la surface elle pourra interagir le polluant.
Les scientifiques ont modifié le gène, exprimant l’une des protéines de la spore d’une bactérie, pour le fusionner avec celui de l’enzyme. Cette fusion permet d’obtenir une protéine avec à la fois les propriétés de celle de la spore et celle de l’enzyme. On parle de protéine de fusion pour désigner une construction à partir de deux protéines. L‘enzyme est ainsi exposée à la surface de la spore. Elle peut ainsi rentrer en contact avec le gaz moutarde présent dans l’environnement.
Vérifier que cette construction fonctionne
Lorsque la fusion des deux protéines a été réalisée, il a fallu vérifier qu’elles étaient capables de dégrader le gaz moutarde. Les scientifiques n’ont pas utilisé directement cette molécule toxique mais à la place une molécule avec des propriétés similaires [10]. Si l’enzyme est bien présente sur la spore, elle dégradera le gaz moutarde ce qui conduit à la libération d’ions chlorure. Ce sont ces ions qui sont recherchés pour estimer la dégradation du polluant. En effet, la concentration est proportionnelle à celle de polluant dégradé.

Les scientifiques indiquent que la spore modifiée, en présence de l’analogue du gaz moutarde, est bien capable de libérer des ions chlorure. Cela signifie que le polluant est dégradé. L’efficacité de cette construction est comparée avec celle de l’enzyme pure. La construction se montre plus résistante que l’enzyme dans des conditions extrêmes telles qu’une température de 60 °C ou de – 20 °C ainsi qu’un pH acide (4) ou basique (10).
Perspectives de l’étude
Cette nouvelle méthode permet de faciliter la production de l’enzyme DhaA tout en augmentant sa résistance à l’environnement. Cela permettra de faciliter le démantèlement d’armes chimiques et le traitement d’autres polluants organochlorés. Le principe d’exposer une protéine à la surface d’une spore est aussi utilisé pour d’autres type de projets telle que la fabrication de vaccin [9].
Référence de l’étude
Wang, F., Song, T., Jiang, H., Pei, C., Huang, Q., & Xi, H. (2019). Bacillus subtilis spore surface display of haloalkane dehalogenase DhaA. Current Microbiology. doi:10.1007/s00284-019-01723-7 (lien)
Pour plus d’informations
[1] Padley, A.P. (2016). Gas: The greatest terror of the Great War. Anaesthesia and Intensive Care, 44(1_suppl), 24–30. doi.org/10.1177/0310057x1604401s05 (lien)
[2] Medvedeva, N., Polyak, Y., Kuzikova, I., Orlova, O., & Zharikov, G. (2008). The effect of mustard gas on the biological activity of soil. Environmental Research, 106(3), 289–295. doi:10.1016/j.envres.2007.0 (lien)
[3] Seventh report of the organisation for the prohibition of chemical weapons – United Nations Joint investigative mechanism (S/2017/904) (lien)
[4] Medvedeva, N., Polyak, Y., Kankaanpää, H., & Zaytseva, T. (2009). Microbial responses to mustard gas dumped in the Baltic Sea. Marine Environmental Research, 68(2), 71–81. doi:10.1016/j.marenvres.2009.04.007 (lien)
[5] Vanninen, P., Östin, A., Bełdowski, J., Pedersen, E. A., Söderström, M., Szubska, M., Grabowski, M., Sieldlewicz, G., Czub, M., Popiel, S., Nawala, J., Dziedzic, D., Jakacki, J., & Pączek, B. (2020). Exposure status of sea-dumped chemical warfare agents in the Baltic Sea. Marine Environmental Research, 105112. doi:10.1016/j.marenvres.2020.105112 (lien)
[6] Prokop, Z., Opluštil, F., DeFrank, J., & Damborský, J. (2006). Enzymes fight chemical weapons. Biotechnology Journal, 1(12), 1370–1380. doi:10.1002/biot.200600166 (lien)
[7] Kulakova, A.N., Larkin, M.J., & Kulakov, L.A. (1997) The plasmid-located haloalkane dehalogenase gene from Rhodococcus rhodochrous NCIMB 13064. Microbiology 143 ( Pt 1):109-115. doi: 10.1099/00221287-143-1-109. (lien)
[8] Curragh, H., Flynn, O., Larkin, M.J., Stafford, T.M., Hamilton, J.T., & Harper, D.B. Haloalkane degradation and assimilation by Rhodococcus rhodochrous NCIMB 13064. Microbiology. 1994;140 (Pt 6):1433‐1442. doi:10.1099/00221287-140-6-1433 (lien)
[9] Ricca, E., & Cutting, S. M. (2003). Emerging applications of bacterial spores in nanobiotechnology. Journal of Nanobiotechnology, 1(1), 6. doi:10.1186/1477-3155-1-6 (lien)
[10] Han, S., Espinoza, L.A., Liao, H., Boulares, A.H., & Smulson, M.E. Protection by antioxidants against toxicity and apoptosis induced by the sulphur mustard analog 2-chloroethylethyl sulphide (CEES) in Jurkat T cells and normal human lymphocytes. British Journal of Pharmacology. 2004 May;142(1):230-230. (lien)








Aucun commentaire